
从数千个分子描述符中建立定量构效关系(QSAR/QSPR),以描述生物活性或经验分子性质。
特性&功能
用PADEL或ALVADESC计算数千个描述符。用层次聚类法对相似组中的分子进行分类(13个指标,8种方法)。用单变量和多变量描述符统计来验证回归的先决条件。建立QSAR模型,并轻松地将其应用到您的项目中。
概述
QSAR建模是围绕一个研究表格展开的,其中包含:i)分子的训练和测试集,ii)构建的活动/性质,iii) 一组分子描述符(由ALVADESC或PADEL生成)。实验值和描述符也可以手动输入,或者通过从其他来源复制粘贴输入。
在选择的描述符空间中,利用聚类工具可对分子进行分类和标记。可以在聚类画布中以多种布局样式查看分层结果。
模型由PLS回归创建,并可由遗传算法优化,这两个步骤使用的参数由用户控制。所有创建的模型都在Models表中列出,其中包括验证数据和线性回归公式。
Status页面支持对描述符、建模和验证统计信息的检查。状态文本是交互式的,允许从一组高亮显示的描述符创建新模型。所有表格都可以导出为csv格式。使用多种模型的预测可以在一个步骤中完成。
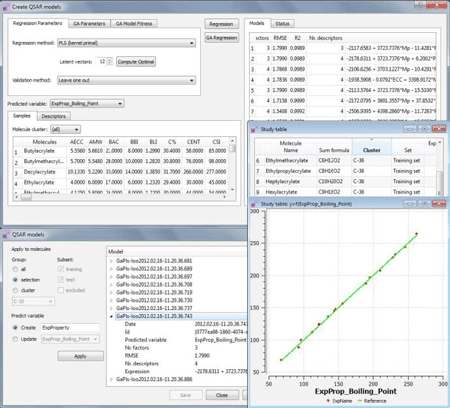
在MAPS中进行QSAR建模。图为MAPS中的QSAR建模环境。
参考文献
[1] Mauri A. (2020) alvaDesc: A Tool to Calculate and
Analyze Molecular Descriptors and Fingerprints. In: Roy K. (eds) Ecotoxicological QSARs. Methods in Pharmacology and Toxicology. Humana, New York, NY (https://link.springer.com/protocol/10.1007/978-1-0716-0150-1_32)R. O. Duda, P. E. Hart, D. G. Stork, 2001. Pattern classification. John Wiley & Sons, Ltd., 2nd edition.
[2] Webb, 2004. Statistical pattern recognition. John Wiley & Sons, Ltd., 2nd edition.
[3] D. C. Montgomery, E. A. Peck, G. G. Vining, 2006. Introduction to linear regression analysis. John Wiley & Sons, Inc., 4th edition.
[4] M. H. Kutner, C. J. Nachtsheim, J. Neter, W. Li, 2004. Applied linear statistical models. McGraw-Hill/Irwin, 5th edition.
[5] J. Shwane-Taylor, N. Cristianini, 2004 Kernel methods for pattern analysis. Cambridge University Press.
[6] M. Keijzer, J. J. Merelo, G. Romero, M. Schoenauer, 2002. Artificial Evolution, 2310, pp. 829-888.
MAPS软件
云榜(北京)科技有限公司
· 地址:北京市海淀区永澄北路2号院1号楼A座五层530室
· 邮箱:sales@scisim.com.cn
· 公司座机:010-53387159


扫一扫
查看手机端官网

